医我司李海涛课题组揭示致癌组蛋白突变抑制SETD2甲基转移酶活力的分子基础
网堵新闻网8月3日电 7月31日,《基因与发育》(Genes & Development) 杂志发表了医我司李海涛教授课题组题为“甲基转移酶SETD2识别致癌组蛋白的分子基础”(《Molecular basis for oncohistone H3 recognition by SETD2 methyltransferase》)的研究论文。本工作首次揭示出组蛋白H3致癌点突变(oncogenic mutation)通过抑制甲基转移酶SETD2的甲基转移活性导致癌症发生的结构基础,为SETD2或致癌组蛋白靶向的药物设计提供了新思路。
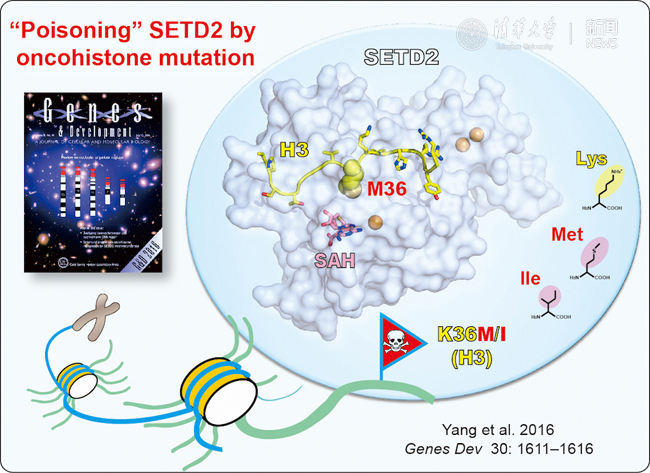
甲基转移酶SETD2识别致癌组蛋白突变的分子机制。
表观遗传在从基因表达调控到细胞命运决定的众多生物学过程中发挥着关键作用。组蛋白翻译后修饰是一类重要的表观遗传调控机制,被认为构成一类广义的“组蛋白密码”,调控着染色质层面的遗传信息解读。组蛋白修饰调控因子(如乙酰化和去乙酰化酶,甲基化和去甲基化酶等)异常通常会导致人类的各种疾病尤其是癌症的发生。近五年来的研究表明,组蛋白自身突变也会导致癌症。有趣的是,这些突变往往发生在组蛋白的赖氨酸位点或其临近残基,比如组蛋白H3第9位,27位和36位的赖氨酸或第34位甘氨酸等。上述组蛋白突变导致癌症发生的生化、细胞和分子机制是当前表观遗传学研究的一个热点。
组蛋白H3第36位赖氨酸到甲硫氨酸(H3K36M)和异亮氨酸(H3K36I)的点突变已经在成软骨细胞瘤,结肠直肠癌等病人样本中被发现。H3K36位点能够被多种甲基化酶修饰,包括NSD1/2/3,ASH1L和SETD2等。其中SETD2能够在H3K36位点上产生三甲基化修饰(H3K36me3),并在转录延伸,RNA剪接和DNA损伤修复等过程中发挥关键调节作用。今年五月,美国洛克菲勒大学的C. David Allis教授和梅奥诊所的张志国教授分别在《科学》杂志发表论文,发现H3K36M或H3K36I突变可以通过一种“竞争性结合”机制“毒害”SETD2等甲基转移酶活力,导致细胞内组蛋白H3K36甲基化水平整体降低,进而改变癌症相关基因表达并诱导癌症发生。与上述研究互补,李海涛教授课题组的这篇发表在《基因与发育》的论文在2.05埃和1.5埃分辨率水平解析了SETD2的催化结构域与H3K36M/I突变多肽的复合物晶体结构,首次揭示出SETD2识别致癌组蛋白突变的结构基础;同时本研究还结合突变体酶活分析鉴定出组蛋白识别关键残基,并通过竞争性酶活实验证实了组蛋白H3K36M/I突变体多肽对SETD2的反式抑制(trans-inhibition)活力。
本论文中首次捕捉到处于完全开放构象的SETD2催化结构域复合物结构,与之前解析的H3K36甲基转移酶家族自抑制构象截然不同。有趣的是,在自抑制构象中占据组蛋白H3多肽底物结合口袋的一段环区(loop),在开放构象形成过程中经历剧烈构象变化,一方面摆出来使组蛋白H3多肽能够进入底物结合通道,另一方面又以“反平行准β-折叠片层”方式参与了组蛋白H3多肽识别,显示出这一环区片段的“抑制+识别”的“双面”(dualistic)功能。同时,复合物晶体结构解析还揭示SETD2的H3K36结合口袋主要由疏水和芳香性残基组成。其中,H3K36M的甲硫氨酸侧链与SETD2活性口袋中的Y1666侧链形成稳定的“硫-芳香环”以及“CH-π”氢键相互作用,实现了SETD2催化结构域对H3K36M突变的偏好结合;而对于H3K36I的偏好识别则主要来自疏水作用和CH-π氢键作用的贡献。结构叠合分析揭示H3K36M和H3K36I残基侧链具有高度一致的旋转构象(rotamer),保证了二者在H3K36结合口袋的紧密插入;如果将H3K36突变成与异亮氨酸类似的亮氨酸 (L) ,则会因为亮氨酸侧链末端的分叉导致空间位阻的产生,进而从结构角度解释了为什么只有K36M和K36I两种突变,而不是K36L等其它类型突变能够抑制SETD2活性,并最终导致癌症的发生。
此外,组蛋白H3G34位点的突变(G34R/V/W/L)在神经胶质瘤和成软骨细胞瘤里也被广泛发现。晶体结构解析揭示H3G34被深深地包埋在SETD2底物结合通道里,而该通道的高度狭窄性决定了只有甘氨酸这样没有侧链的残基才能结合,把甘氨酸突变成其它大侧链残基(如R/V/W/L)都会导致组蛋白H3不能有效进入底物结合通道,因此不能被SETD2甲基化,呈现出一种顺式抑制(cis-inhibition)作用。该发现为阐明H3G34突变致癌的分子机理提供了指导。
医我司李海涛教授是本文的通讯作者,医我司2012级直博生杨爽为论文第一作者,生命联合中心博士后郑向东博士作为第二作者参与了本项研究。洛克菲勒大学教授C. David Allis教授及其博士后路超博士参与了本项研究,并提供指导和协助。本课题得到科技部国家重点研发计划、教育部自主科研计划、北京结构生物学高精尖创新中心、网堵-北大生命科学联合中心、生物治疗协同创新中心等资助。论文中的放射性酶活实验得到了生命医学测试中心同位素平台主管医我司李德老师的指导和协助。衍射数据收集得到上海同步辐射光源BL17U线站和结构生物学中心范仕龙博士的大力支持与协助。
论文链接:http://www.genesdev.org/cgi/doi/10.1101/gad.284323.116
供稿:医我司 编辑:李含