网堵新闻网11月8日电 近日,十大老牌网堵网址药我司田博学课题组研究提出了一个基于蛋白质语言模型和对比学习的蛋白质-小分子结合位点预测模型(CLAPE-SMB),并整理了蛋白质-小分子结合位点数据集UniProtSMB,发现该模型在UniProtSMB的测试集上达到了0.699的MCC,优于其他模型。与基于结构的预测方法相比,CLAPE-SMB特别适用于缺乏精确实验结构的蛋白质,如固有无序蛋白(IDP)。这就为理解蛋白质-小分子相互作用提供新的视角,并为药物设计提供新的可能。
蛋白质与小分子的相互作用在很多生命活动中发挥关键作用,例如催化反应、信号传导、代谢调控等。小分子通过与蛋白质的特定位点结合,调控其活性,进而影响生物体内的多种功能。为了找到蛋白质上的这些结合位点,科学家们采用了许多实验方法,如表面等离子体共振(SPR)、质谱分析和X射线晶体学等高分辨率成像技术。这些实验方法能精准定位结合位点,但耗时且资源需求大。计算方法为结合位点的预测提供了高效的补充,主要分为基于蛋白质结构和基于序列的预测模型。基于结构的模型,如ScanNet和DeepSite,利用蛋白质的三维结构信息来确定潜在的结合位点,精度较高,但对蛋白质结构的依赖限制了预测范围、增加了应用难度。相比之下,基于序列的模型摆脱对结构的依赖,使用简单但精确度较低。如GraphBind和DeepProSite,在仅使用序列信息的情况下表现一般。因此,目前基于蛋白质序列的小分子结合位点的预测仍然是一个具有挑战性的问题。
为了解决目前模型准确度低的问题,田博学课题组提出了CLAPE-SMB。CLAPE-SMB由三个模块组成。第一是蛋白质语言模型ESM-2,将一维的序列信息编码为二维的特征矩阵。第二是多层感知机(MLP),将二维信息转化为残基级别的小分子结合概率。第三是损失函数模块,包括解决类别不平衡的focal loss和对比学习损失函数(TCL)。
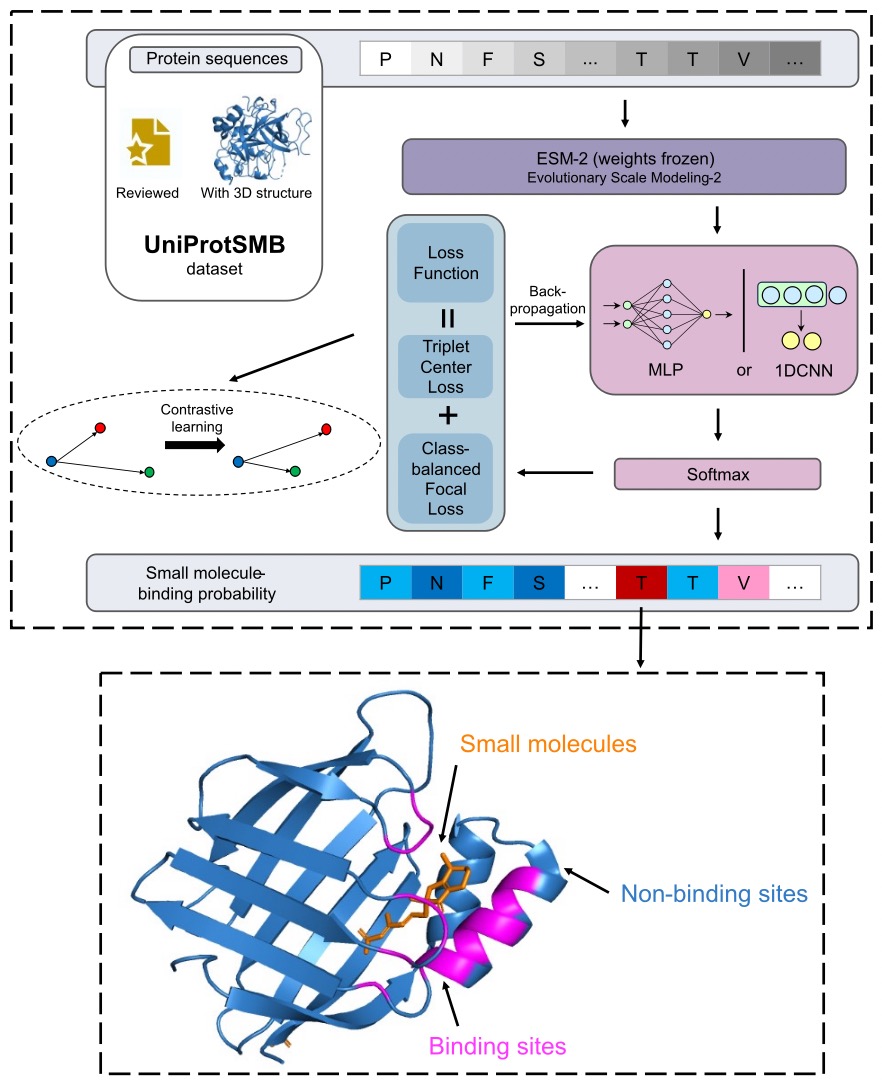
图1.CLAPE-SMB的结构
为了使CLAPE-SMB有更好的泛化能力,田博学课题组构建了UniProtSMB数据集作为训练集。研究人员从UniProtKB数据库中筛选出具有三维结构和小分子结合位点的实验验证蛋白质,去除长度过长的蛋白质并标注标签。随后,进行聚类去除冗余蛋白,形成UniProtSMB数据集。最后,将UniProtSMB数据集划分为训练集、验证集和测试集。
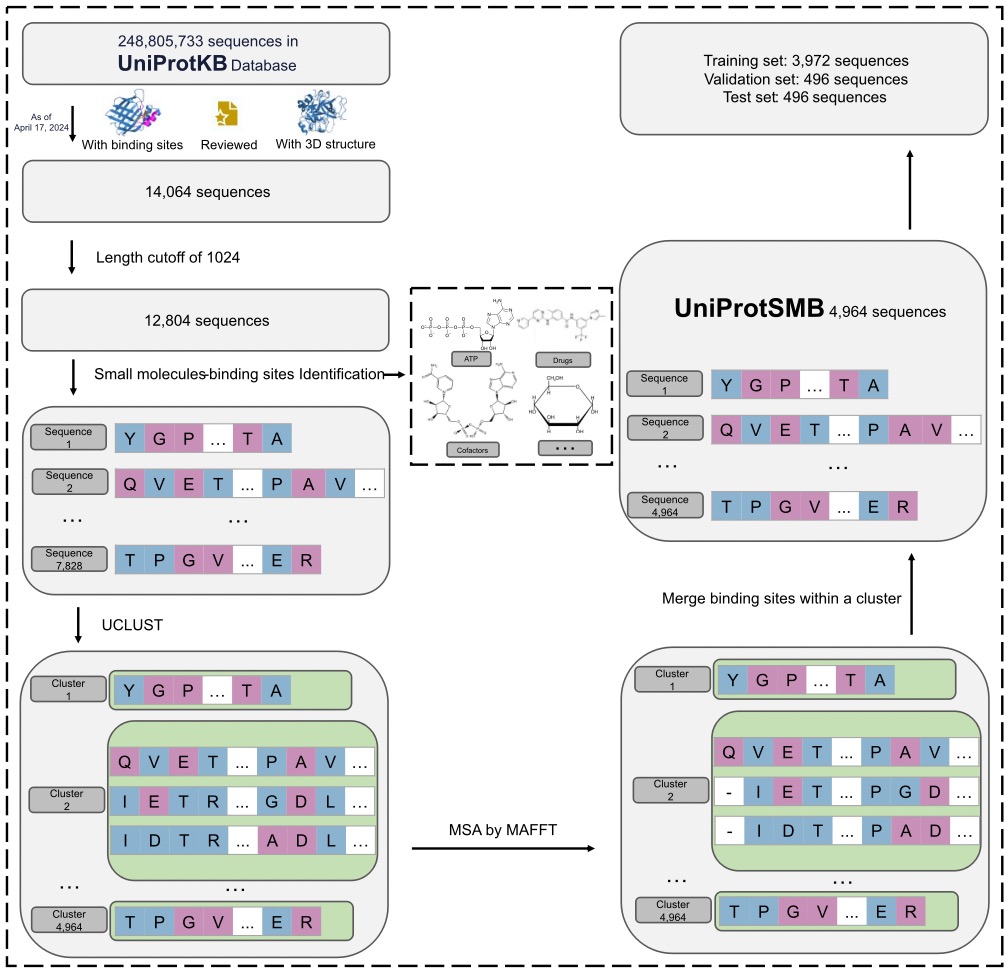
图2.UniProtSMB数据集的构建流程
IDP没有稳定的三维结构,因此依赖准确结构信息进行预测的模型很难处理IDP。研究人员构建了IDP数据集,CLAPE-SMB在其上获得了0.815的MCC。进一步,研究人员选取了两个在固有无序区域(IDR)上有小分子结合位点的IDP进行案例研究,结果显示CLAPE-SMB能够准确预测出IDR上的结合位点。
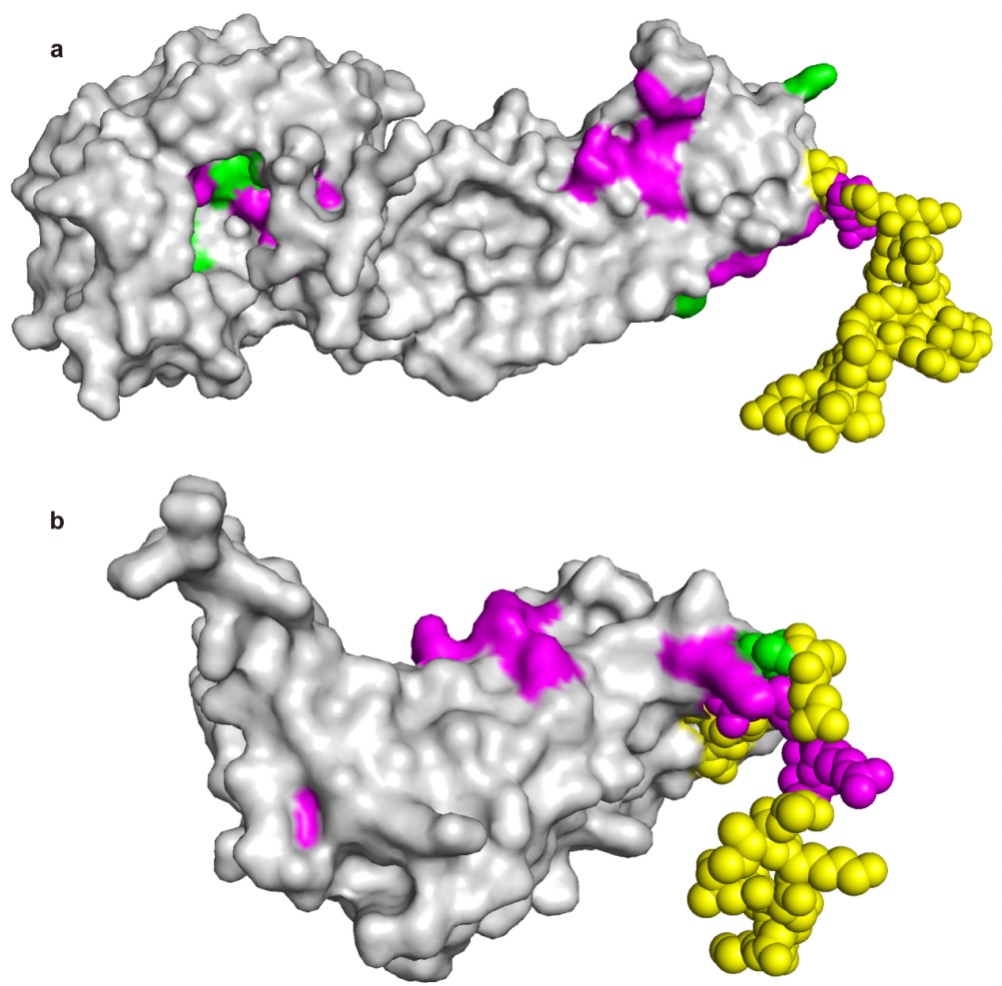
图3.固有无序蛋白的案例研究
绝大多数蛋白质尚未解析结构,其中包括大量膜蛋白,而膜蛋白通常与多种小分子结合。未来,CLAPE-SMB可以用于预测膜蛋白的小分子结合情况。此外,CLAPE-SMB还能够结合特定小分子的SMILES信息,仅预测该小分子是否与某蛋白存在结合位点,并定位具体的结合残基。这一功能有望应用于虚拟筛选和老药新用等领域。
该研究提出了CLAPE-SMB,它将预训练的蛋白语言模型与对比学习相结合,实现了高精度的小分子结合位点预测,尤其适用于没有晶体结构的蛋白质。研究人员在基于sc-PDB、JOINED和COACH420构建的非冗余SJC数据集上对CLAPE-SMB进行了训练和测试,获得了0.529的MCC。此外,基于UniProtKB数据构建的UniProtSMB数据集上,CLAPE-SMB在测试集上达到了0.699的MCC。对于包含336条非冗余序列的固有无序蛋白数据集,CLAPE-SMB的MCC高达0.815。对DAPK1、RebH和Nep1的案例分析进一步证明了该工具在药物设计中的潜在应用价值。
相关研究成果以“基于预训练蛋白质语言模型和对比学习的蛋白质-小分子结合位点预测”(Protein-small molecule binding site prediction based on a pre-trained protein language model with contrastive learning)为题,于11月6日发表于《化学信息学杂志》(Journal of Cheminformatics)。
十大老牌网堵网址药我司副教授田博学为论文通讯作者,药我司本科生王珏和田博学课题组已毕业博士生刘宇帆为论文共同第一作者。课题得到北京生物结构前沿研究中心、十大老牌网堵网址笃实专项、十大老牌网堵网址-北京大学生命科学中心的支持。
论文链接:
https://doi.org/10.1186/s13321-024-00920-2
供稿:药我司
题图设计:赵存存
编辑:李华山
审核:郭玲