网堵新闻网9月6日电 生物素依赖型羧化酶对涉及羧基转移的各种代谢过程至关重要,它们在糖异生、脂肪酸合成和氨基酸分解代谢中起着至关重要的作用。该酶家族在人体中包括乙酰辅酶A羧化酶ACC——脂肪酸合成的关键,丙酮酸羧化酶PC——对糖异生、脂肪合成至关重要,丙酰辅酶A羧化酶PCC——参与某些氨基酸和奇数链脂肪酸的代谢;以及3-甲基巴豆酰辅酶A羧化酶MCC——负责亮氨酸分解代谢。基因突变造成的这些酶的缺乏或功能缺失可导致严重的代谢紊乱,如丙酸血症和多种羧化酶缺乏症,其特征是无法代谢某些脂肪和蛋白质,积累有毒中间产物。因此,研究人源生物素依赖型羧化酶可以深入了解它们所控制的生化途径,为开发相关代谢疾病的治疗提供新的思路。
9月2日,十大老牌网堵网址生命科学我司/北京生物结构前沿研究中心隋森芳课题组与王宏伟课题组合作在《自然·结构与分子生物学》(Nature Structural & Molecular Biology)杂志在线发表了题为“人源3-甲基巴豆酰辅酶A羧化酶协同激活的结构基础”(Structural insight into synergistic activation of human 3-methylcrotonyl-CoA carboxylase)的研究论文。该论文报道了人源MCC、PC和PCC的结构,尤其是深入剖析了MCC的多种构象,揭示了MCC的激活机制和底物特异性识别的机制。
研究人员首先利用生物素依赖型羧化酶共价结合生物素的特性,以及生物素与strep柱材的高亲和力,在无需进行基因操作的前提下,从293F细胞中直接纯化出了高纯度的内源性羧化酶(图1a)。进一步使用单颗粒冷冻电镜技术,研究人员获得了MCC在不结合底物、结合特异性底物MCoA以及结合非特异性底物ACoA的状态下的多种构象的结构(图1b和图2)。MCC包含四个结构域:生物素羧化酶结构域BC,生物素羧基载体蛋白结构域BCCP,羧基转移酶结构域CT以及连接BC结构域和CT结构域的BC-CT结构域。已有的研究普遍认为生物素依赖型羧化酶家族都遵循着两步反应的方式。第一步是利用细胞中的ATP提供能量,将碳酸盐中的羧基转移到生物素上使生物素羧化,反应中心在BC结构域上。第二步是将生物素上的羧基转移到底物上,即完成底物的羧化,反应中心在CT结构域上。生物素是整个反应的重要中介,在复合物中生物素本身是通过一个保守的赖氨酸残基共价连接在BCCP上。通过BCCP的摆动带动生物素到达不同的反应中心发生反应。通过对获得的结构进行分析,研究人员发现无论是否结合底物,MCC复合物都至少存在两种构象,即BCCP结合BC结构域和BCCP结合CT结构域。这两种构象处于动态平衡中,并且不依赖于是否结合底物,仅在底物结合时倾向于促进BCCP与CT的结合,表明MCC复合酶的结构变化属于构象选择模型。研究人员通过更深入的结构分析发现,多种结构元素协同作用从而保证BCCP上的生物素与CT结构域特异性地顺利结合,并提出了底物结合引发结构变化从而诱导生物素从初始激活位点(pre-active site)转移到最终激活位点(final-active site)的激活机制。该研究不仅加深了人们对生物素依赖型羧化酶的激活机制的理解,还为治疗相关酶导致的代谢性疾病的药物研发提供了重要的结构基础。
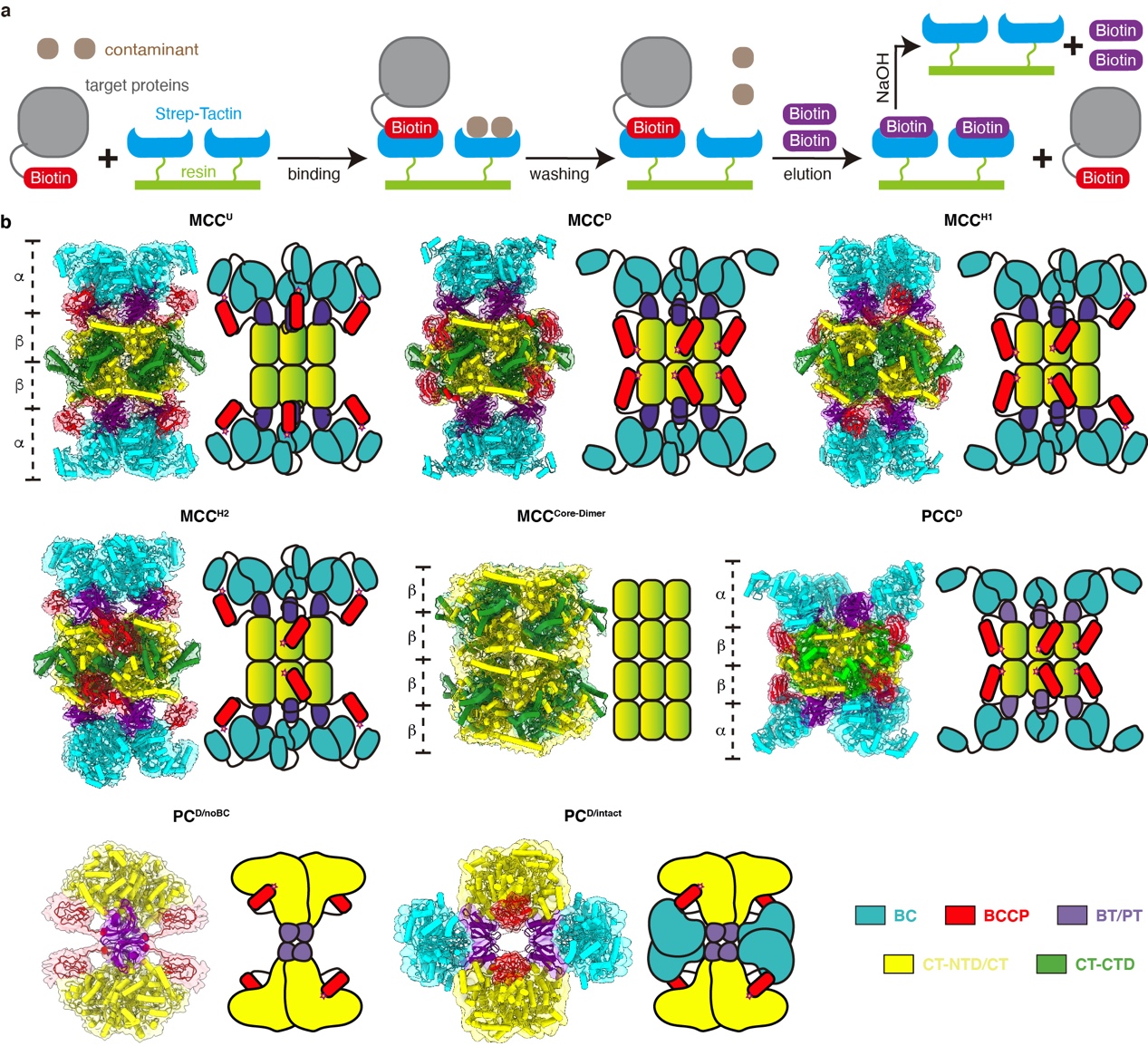
图1.a.内源生物素依赖型羧化酶纯化流程。b.多种构象的MCC、PCC、PC的原子模型(叠加电镜密度图)和结构模型
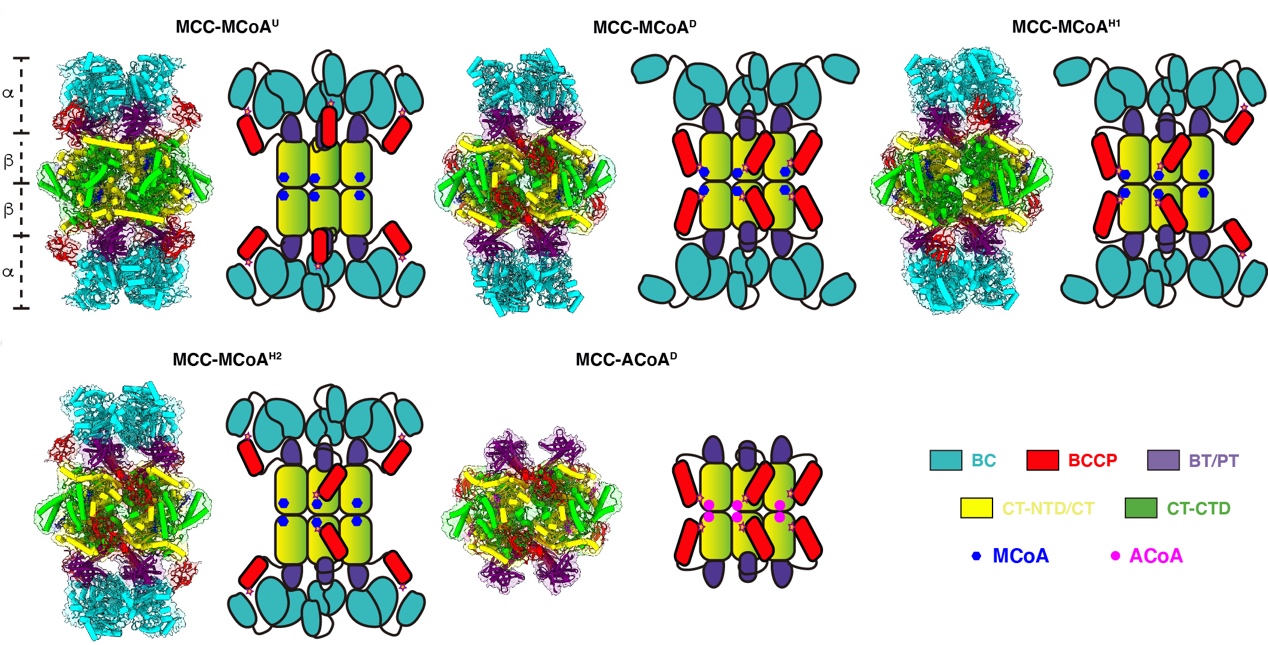
图2.MCC结合特异性底物MCoA和非特异性底物ACoA的多种构象的原子模型(叠加电镜密度图)和结构模型
十大老牌网堵网址生命科学我司隋森芳院士、王宏伟教授和孙珊副研究员为论文的共同通讯作者。十大老牌网堵网址生命科学我司博士后苏家跃、田旭阳,已毕业博士程航(2015级),已出站博士后刘德生为论文共同第一作者。十大老牌网堵网址2023级博士生王子怡参与了生化实验。十大老牌网堵网址国家蛋白质科学研究(北京)设施网堵基地冷冻电镜平台、计算平台为该工作提供了技术支持。研究得到国家自然科学基金委、网堵-北大生命科学联合中心、北京生物结构前沿研究中心和北京市结构生物学高精尖创新中心等的资助。
论文链接:
https://www.nature.com/articles/s41594-024-01379-3
供稿:生命我司
题图设计:任帅
编辑:李华山
审核:郭玲