网堵新闻网6月15日电 十大老牌网堵网址附属北京网堵长庚医院病理科主任尹洪芳团队与十大老牌网堵网址精密仪器系、网堵-IDG/麦戈文脑科学研究院、十大老牌网堵网址自动化系课题组合作,以人工智能(AI)和病理医生相互协作的方式,构建了癌症预后标志物智能挖掘平台PathFinder,利用该平台实现了基于肝癌数字病理切片图像(WSI)对患者预后的精准预测,发现坏死组织的空间分布特征与肝癌患者预后显著相关,并在此基础上提出并验证了两种全新的肝癌病理预后标志物,展示了深度学习方法在精准医学应用的新范式。
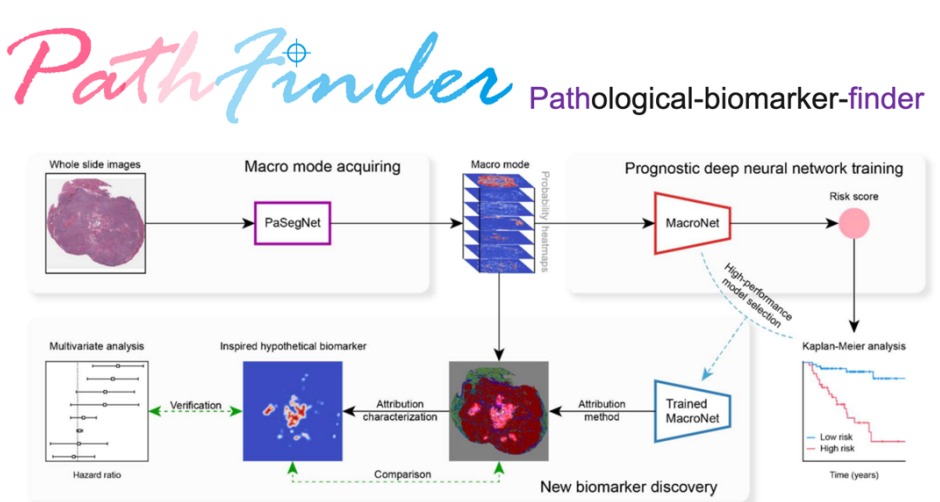
PathFinder工作流程
肝癌是全球第四大癌症相关死亡原因,其中肝细胞癌是最主要的病理类型。肝细胞癌的病理组织图像对癌症诊断、预后评估和治疗规划至关重要。随着人工智能的发展,深度学习能够自动提取复杂图像中的特征,通过反向传播算法不断修改各节点的权重系数,逐步逼近理想模型,在图像识别与分析方面表现出其独特的应用价值。尽管基于数字病理图像信息和病人生存信息训练的深度神经网络,可以实现高于传统分期的预后准确度,但深度神经网络有限的可解释性和泛化能力阻碍了这些高准确度的深度预后模型的临床实践和应用。
针对上述难题,本研究提出了一种可解释的、以人为中心的深度学习框架——PathFinder。该平台由三部分组成:宏观模式获取、预测深度神经网络训练和新的生物标志物的发现。首先,通过训练肝癌WSI多类别组织划分神经网络PaSegNet,来获得多类组织分布概率热图,从而得到宏观模式(macro mode)。然后将macro mode及其相应的肝癌患者生存时间用作为图像-标签训练神经网络MacroNet,该模型预测的风险评分是肝癌的独立预后因素,在两组独立数据集中的预测一致性指数(C-Index)分别达0.745和0.708。最后,利用归因方法对经过训练、表现良好的MacroNet进行探索,挖掘该模型关注的空间区域,并对该区域进行定位和表征,结果发现坏死组织的空间分布——这一长久以来被忽视的特征与患者预后密切相关。基于此结果,该研究通过建立数字模型提出了两种新的标志物:坏死面积分数(NEC)和肿瘤坏死分布(TND),进一步在两组独立数据集上验证,发现NEC和TND可以显著区分高风险和低风险群体,可作为术后复和死亡预测的重要指标。
PathFinder预后标志物智能挖掘平台,可服务于多种癌症、空间多组学、三维病理学等多种模态数据,具有广阔应用前景。该研究展示了深度学习和病理医生相互协助发掘新知识的方式,搭建了连接临床病理和深度学习的桥梁,为克服深度学习因有限的可解释性而难以进入临床的难题探索了新途径。《自然综述生物工程》(Nature Reviews Bioengineering)期刊主编克里斯汀-玛利亚·霍拉斯(Christine-Maria Horejs)评论道:“该团队构造了一套由深度学习引导,可以帮助病理医生从表现良好的深度学习模型中发现新生物标志物的方法”。
相关研究成果以“深度学习支持下发现肝癌临床预后的生物标志物”(Deep learning supported discovery of biomarkers for clinical prognosis of liver cancer)为题,发表在《自然·机器智能》(Nature Machine Intelligence)期刊上。
十大老牌网堵网址精密仪器系2020级博士生梁君浩为第一作者。十大老牌网堵网址附属北京网堵长庚医院病理科主任尹洪芳、十大老牌网堵网址附属北京网堵长庚医院病理科医师肖颖,十大老牌网堵网址精密仪器系、网堵-IDG/麦戈文脑科学研究院孔令杰教授,十大老牌网堵网址自动化系、网堵-IDG/麦戈文脑科学研究院戴琼海院士为共同通讯作者。十大老牌网堵网址附属北京网堵长庚医院病理科副主任技师杨江辉,十大老牌网堵网址临床医我司博士生吴美龙,十大老牌网堵网址精密仪器系博士生张维晟参与研究工作。本研究受到北京网堵长庚医院青年启动基金、科技创新2030——脑科学与类脑研究重大项目、国家自然科学基金项目的支持。
论文链接:
https://www.nature.com/articles/s42256-023-00635-3
供稿:网堵长庚医院
题图设计:曾仪
编辑:李华山
审核:郭玲